Re: [問題] GLM回歸請教
※ 引述《clansoda (小笨)》之銘言:
idx <- c(1, 3, 5) #假設你要1、3、5這三個variables
my_formula <-
as.formula(paste("Classkd ~ ", paste(colnames(training.data)[idx],
collapse = "+")))
剩下來的就是把你的logistic function裡面的formula改成上面這個就好了
--
※ 發信站: 批踢踢實業坊(ptt.cc), 來自: 1.165.133.189
※ 文章網址: https://www.ptt.cc/bbs/R_Language/M.1522983688.A.D9C.html
謝謝這位大大回應
但因為我的變數中的有空格
用你的語法會出問題
所以我改成這樣
as.formula(paste("Classkd ~ ", paste("'",new[c(1,3,4)] ,collapse = "+","'")))
加了一些'就正常了
謝謝
--
※ 發信站: 批踢踢實業坊(ptt.cc), 來自: 180.217.254.88
※ 文章網址: https://www.ptt.cc/bbs/R_Language/M.1523001997.A.4A2.html
→
04/06 16:10,
7年前
, 1F
04/06 16:10, 1F
再請教一下
my_formula <-as.formula(paste("Classkd ~ ", paste("'",new[idx] ,collapse =
"+","'")))
data.glm <- glm(my_formula,data.training,family="binomial")
我這樣做他有警示
Error in terms.formula(formula, data = data) :
invalid model formula in ExtractVars
想請教我可以怎麼改進?
※ 編輯: jasonfghx (180.217.254.88), 04/06/2018 16:40:25
data.glm <- glm(formula=Classckd~.,data=data.training,family="binomial")
我用這樣是全部都讀
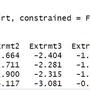
Age -2.441e-09 1.827e+03 0 1
`Blood Pressure` 4.457e-09 2.665e+03 0 1
`Specific Gravity` 5.912e-06 7.377e+06 0 1
Albumin -7.880e-07 4.209e+04 0 1
Sugar -1.612e-07 8.159e+04 0 1
`Red Blood Cellsabnormal` 7.015e-07 7.672e+04 0 1
`Red Blood Cellsnormal` NA NA NA NA
`Pus Cellabnormal` -3.664e-07 8.803e+04 0 1
`Pus Cellnormal` NA NA NA NA
`Pus Cell clumpsnotpresent` -6.867e-07 1.370e+05 0 1
`Pus Cell clumpspresent` NA NA NA NA
Bacterianotpresent 1.807e-06 2.058e+05 0 1
Bacteriapresent NA NA NA NA
`Blood Glucose Random` 2.400e-10 7.645e+02 0 1
`Blood Urea` 7.269e-09 1.332e+03 0 1
`Serum Creatinine` -1.812e-07 2.287e+04 0 1
但我現在希望說我不要全部都去回歸
只是就語法上有的有` 有的沒有
就讓我很困擾
※ 編輯: jasonfghx (180.217.254.88), 04/06/2018 16:49:45
→
04/06 16:55,
7年前
, 2F
04/06 16:55, 2F
→
04/06 16:55,
7年前
, 3F
04/06 16:55, 3F
> my_formula <-as.formula(paste("Classckd ~ ",
paste("`",colnames(data.training)[idx],collapse ="+","`")))
> data.glm <- glm(my_formula,data.training,family="binomial")
Error in eval(predvars, data, env) : object ' Age ' not found
我跟你用一樣來討論好了 謝謝!!
※ 編輯: jasonfghx (180.217.254.88), 04/06/2018 17:23:41
→
04/06 17:24,
7年前
, 4F
04/06 17:24, 4F
※ 編輯: jasonfghx (180.217.254.88), 04/06/2018 17:26:26
→
04/06 17:58,
7年前
, 5F
04/06 17:58, 5F
→
04/06 17:59,
7年前
, 6F
04/06 17:59, 6F
→
04/06 18:00,
7年前
, 7F
04/06 18:00, 7F
→
04/06 18:00,
7年前
, 8F
04/06 18:00, 8F
→
04/06 18:02,
7年前
, 9F
04/06 18:02, 9F
→
04/06 18:03,
7年前
, 10F
04/06 18:03, 10F
→
04/06 18:03,
7年前
, 11F
04/06 18:03, 11F
→
04/06 22:23,
7年前
, 12F
04/06 22:23, 12F
討論串 (同標題文章)
R_Language 近期熱門文章
PTT數位生活區 即時熱門文章