[問題] 如何一次檢查多個column的字串
[問題類型]:
程式諮詢(我想用R 做某件事情,但是我不知道要怎麼用R 寫出來)
[軟體熟悉度]:
入門(寫過其他程式,只是對語法不熟悉)
[問題敘述]:
代碼儲存在複數欄位中,希望檢查是否有某些欄位符合特定字串規則,輸出True or False
目前用 dplyr::filter_at 是可以得到需要的結果
但需要另外產生一個data.frame再join回來
且要檢查多組不同字串規則時變得更為冗長
想問看看有沒有更好的寫法
ex:
ID 代碼一 代碼二 ... 代碼N
1 O33 O34 O354
2 O331 O354
3 OO33 O345
需要的結果
ID 代碼一 代碼二 ... 代碼N O33開頭 O34開頭 ...
1 O33 O34 O354 True True
2 O331 O354 True False
3 OO33 O345 False True
[程式範例]:
df <- read.csv( ...... )
require(dplyr)
dftemp <- filter_at(df, vars(starts_with("代碼")), any_vars(grepl("^O33", .)))
dftemp$O33開頭 <- True
df <- left_join(df, dftemp )
df$O33開頭 <- ifelse( is.na(df$O33開頭), False, True)
然後為 "O34開頭" 再做一次...
--
So stand by your glasses steady,
Here’s good luck to the man in the sky,
Here’s a toast to the dead already,
Three cheers for the next man to die.
--
※ 發信站: 批踢踢實業坊(ptt.cc), 來自: 118.163.222.252 (臺灣)
※ 文章網址: https://www.ptt.cc/bbs/R_Language/M.1599453865.A.4D5.html
※ 編輯: daze (118.163.222.252 臺灣), 09/07/2020 12:54:34
→
09/07 14:06,
5年前
, 1F
09/07 14:06, 1F
代碼1 ~ 代碼N 都可能出現O33開頭的值
N的數量不固定
用 filter_at 可以抓 column name 裡面有"代碼"開頭的columns
另外還可能有其他開頭不是"代碼"的columns
這些columns不可列入檢查
→
09/07 17:15,
5年前
, 2F
09/07 17:15, 2F
→
09/07 17:15,
5年前
, 3F
09/07 17:15, 3F
→
09/07 17:23,
5年前
, 4F
09/07 17:23, 4F
這個code只在代碼1比對 O33, 在代碼2比對 O34。
在代碼2出現O33就不行了。
當然寫成巢狀 for loop也可以
但是這樣實現的話
使用 filter_at 的簡潔度與可讀性可能更好些
而且在N會變動的情況下
巢狀 for loop 會更麻煩些
※ 編輯: daze (114.40.18.158 臺灣), 09/07/2020 18:14:49
→
09/07 18:35,
5年前
, 5F
09/07 18:35, 5F
→
09/07 18:36,
5年前
, 6F
09/07 18:36, 6F
推
09/08 13:42,
5年前
, 7F
09/08 13:42, 7F
→
09/08 13:42,
5年前
, 8F
09/08 13:42, 8F
更之前有試著用 reshape2::melt/dcast
行為跟 tidyr::gather/spread 應該很類似
不過有個問題是 melt 後 dataframe 會變大好幾倍
比較小的檔案可以這麼做,但6G的檔案變大10倍,記憶體就爆了
當時的做法是把某些暫時不用的 column 抽掉先把檔案瘦身
跑完 melt/dcast 之後再 join 回去
但這樣當然比較麻煩,又容易出錯
目前處理的檔案大小,最大就 10G 左右
剛好落在用 filter_at 不會爆而用 melt 就會爆的狀態
filter_at 在效能上對我來說其實剛好夠用,能用的參數也算方便
但 filter_at 為了 filter,應該本來就會為每一 row 產生一個邏輯值
我主要的困擾是
希望有現成的類似函式可以直接產生該邏輯值寫入新 column
而不必使用 filter_at 生成新 dataframe 再 join 回去
從而可以直接對 patterns list 做 lapply
※ 編輯: daze (114.40.18.158 臺灣), 09/08/2020 16:53:57
做兩次 lapply 再 cbind,最後把 column name 改掉。
缺點是可能過兩個月我就搞不清這段code的作用了。
require(dplyr)
df<- read.csv(...)
pattern.list <- c("^O33", "^O34")
CNames<-colnames(df)
df<-df %>% cbind( lapply( pattern.list, function(y) Reduce("|",
lapply(select(.,starts_with("代碼")), function(x) grepl(y, x)))))
colnames(df)<-c(CNames,pattern.list)
※ 編輯: daze (114.40.18.158 臺灣), 09/08/2020 21:56:34
這樣似乎稍微好一點...
cbind(df, lapply(pattern.list, function(x) df %>% rowwise() %>% summarise(
(sum(grepl( x, across(starts_with("代碼"))))>0))))
但這樣不是非常有效率
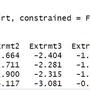
跑 1.7M rows, 在 6個 column中比對1個pattern, 大概要 3~4 分鐘
用一開始的 filter_at 實現同樣的比對則要花... 3 秒鐘。
※ 編輯: daze (114.40.18.158 臺灣), 09/08/2020 23:32:04
推
09/09 09:36,
5年前
, 9F
09/09 09:36, 9F
推
09/09 20:06,
5年前
, 10F
09/09 20:06, 10F
→
09/09 20:06,
5年前
, 11F
09/09 20:06, 11F
試著讀了一個約 5G 的 CSV 檔進來 (12568899 rows x 43 columns)
Rstudio 顯示 data.frame 大小是 6259133200 bytes
將 6 個代碼欄 gather 成一個
data.frame 大小變成 22696429616 bytes
在工作管理員中, Rsession.exe 的記憶體用量則約 28G
電腦開始 lag, 畢竟實體記憶體只有 32G
把 R 重啟後,用 melt 做同樣的操作
data.frame 大小變成 22394776472 bytes
電腦仍然開始 lag
所謂 gather 比 melt 好很多,具體是指哪方面?
※ 編輯: daze (36.237.70.94 臺灣), 09/09/2020 23:24:29
推
09/11 12:28,
5年前
, 12F
09/11 12:28, 12F
→
09/11 12:34,
5年前
, 13F
09/11 12:34, 13F
→
09/11 12:36,
5年前
, 14F
09/11 12:36, 14F
→
09/11 12:38,
5年前
, 15F
09/11 12:38, 15F
※ 編輯: daze (114.39.55.253 臺灣), 09/13/2020 22:00:23
※ 編輯: daze (114.39.55.253 臺灣), 09/14/2020 23:53:52
R_Language 近期熱門文章
PTT數位生活區 即時熱門文章