[問題] 用 R 做卡方檢測
[問題類型]: 程式諮詢
[軟體熟悉度]: 入門
主要用 perl 處理資料,偶爾用R作分析
[問題敘述]:
我現在有兩筆資料要比較其組成比例有沒有差異
問過其他人後知道要用卡方
資料大致上長這樣
要比較 A、T、C、G 的組成在 A 跟 B 之間是否有差異
A T C G
SampleA 1937 373 14788 238
sampleB 2590 395 17686 274
想請問一下要怎麼在R上面做到卡方檢定?
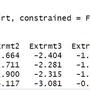
目前 google 查到的方法是這樣
a <- c(1937,373,14788,238)
b <- c(2590,395,17686,274)
chisq.test(rbind(a,b))
在上面列出來case時結果跟我在網路上找到的計算器結果相同
(http://www.socscistatistics.com/tests/chisquare2/Default2.aspx)
可是到了另一個 case
A T C G
SampleA 1595826 308 450 76
sampleB 1576617 362 428 59
上面的寫法跑出來的p-value是 0.06327
可是計算器上給的結果是 The p-value is < 0.00001
想請問一下正確的做法是怎麼樣
[環境敘述]:
R version 3.3.2 (2016-10-31)
Platform: x86_64-redhat-linux-gnu (64-bit)
Running under: CentOS release 6.7 (Final)
locale:
[1] C
attached base packages:
[1] stats graphics grDevices utils datasets methods base
--
※ 發信站: 批踢踢實業坊(ptt.cc), 來自: 118.163.96.158
※ 文章網址: https://www.ptt.cc/bbs/R_Language/M.1492571058.A.9D8.html
推
04/19 11:19, , 1F
04/19 11:19, 1F
推
04/19 11:58, , 2F
04/19 11:58, 2F
推
04/21 09:39, , 3F
04/21 09:39, 3F
推
04/21 12:14, , 4F
04/21 12:14, 4F
R_Language 近期熱門文章
PTT數位生活區 即時熱門文章