[問題] 以helmert coding做dummy variables給RDA
[問題類型]:
程式諮詢(我想用R 做某件事情,但是我不知道要怎麼用R 寫出來)
query ?model.matrix 用contr.helmert來處理類別變數,所得到的dummy variables
應該是彼此正交 (orthogonal) 但試驗過程中不必然如此,以?model.matrix 提供的例
子的確是得到正交的結果,但下方範例則未必(只有tb接近,ta, tc則沒有)
不知道是不是自己哪裡搞錯?? 剛去翻Numerical ecology with R 書中也是這樣處理
dummy variables, 只是沒提到model.matrix出來的結果是否彼此正交
一般的連續變數彼此相關,但還是可以做為RDA(比如vegan::rda())的解釋變數,但
dummy variables如果彼此存在相關性,不知道是否適當?...
請教各位,謝謝!
[程式範例]:
library(data.table)
dta <- fread('
2 2 2 3 3 3 3 3 4 4 4 4 4 4 4 4 4 4 5 4
4 4 4 4 2 2 2 3 3 3 3 4 4 4 4 4 4 4 4 4
4 4 5 4 4 4 5 5 5 5 5 5 4 4 4 4 3 3 3 3
3 2 2 2 2 2 2 2 2 1 1 1 5 5 5 4 4 4 3 3
3 3 3 3 2 2 2 2 2 2 2 1 1 1 1 1 5 4 4 4') %>% unlist(use.names = F)
dtb <- fread('
3 4 4 2 1 2 1 4 2 5 3 4 2 2 2 1 2 5 1 2
1 3 2 4 3 3 1 1 4 4 2 5 5 3 2 1 4 5 4 5
4 1 4 2 1 1 2 1 2 3 4 3 3 5 2 1 1 5 1 4
2 1 3 2 4 1 3 4 1 5 4 2 4 3 3 1 5 1 2 2
3 5 2 2 1 1 2 4 1 5 5 1 5 4 4 3 4 4 3 3') %>% unlist(use.names = F)
data <- data.frame(tsta=as.factor(dta),
tstb=as.factor(dtb),
tstc=as.factor(sample(1:5,100,replace=T)))
ta <- model.matrix(~tsta, data, contrasts = list(tsta = "contr.helmert"))
cor(ta[,-1]) ########################################## collinear results
###### tsta1 tsta2 tsta3 tsta4
#tsta1 1.00000000 -0.26595995 -0.22219661 -0.09186113
#tsta2 -0.26595995 1.00000000 -0.09463643 -0.03912485
#tsta3 -0.22219661 -0.09463643 1.00000000 -0.13350694
#tsta4 -0.09186113 -0.03912485 -0.13350694 1.00000000
tb <- model.matrix(~tstb, data, contrasts = list(tstb = "contr.helmert"))
cor(tb[,-1])##################################### almost orthogonal
###### tstb1 tstb2 tstb3 tstb4
#tstb1 1.000000000 0.01192291 0.009193480 0.005885889
#tstb2 0.011922914 1.00000000 0.076964759 0.049274704
#tstb3 0.009193480 0.07696476 1.000000000 -0.004985713
#tstb4 0.005885889 0.04927470 -0.004985713 1.000000000
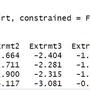
tc <- model.matrix(~tstc, data, contrasts = list(tstc = "contr.helmert"))
cor(tc[,-1]) ##################################### collinear
###### tstc1 tstc2 tstc3 tstc4
#tstc1 1.00000000 0.14295322 0.10340384 0.07009347
#tstc2 0.14295322 1.00000000 -0.03144958 -0.02131846
#tstc3 0.10340384 -0.03144958 1.00000000 -0.03976866
#tstc4 0.07009347 -0.02131846 -0.03976866 1.00000000
tt <- model.matrix(~tsta, data)
cor(tt[,-1]) ### binary coding produce also non-orthogonal results
[關鍵字]:
選擇性,也許未來有用
--
※ 發信站: 批踢踢實業坊(ptt.cc), 來自: 140.112.65.48
※ 文章網址: https://www.ptt.cc/bbs/R_Language/M.1475569846.A.5BE.html
→
10/04 17:18, , 1F
10/04 17:18, 1F
→
10/04 17:18, , 2F
10/04 17:18, 2F
→
10/04 17:19, , 3F
10/04 17:19, 3F
→
10/04 17:38, , 4F
10/04 17:38, 4F
R_Language 近期熱門文章
PTT數位生活區 即時熱門文章