[分享] bigmemory 套件分享
1. 套件名稱:
bigmemory 4.5.19
2. 套件主要用途:
處理較吃記憶體的資料,尤其當資料大小逼近或超過實體記憶體,使load資料很慢時..
但資料限定為單一資料型態,不能同時混雜character, numeric
它這種資料結構 big.matrix 其實只是一個 R object但實際指向 C++資料結構的指標
可以memory或檔案形式 share (shared.big.matrix, filebacked.big.matrix),實現
在 multip processes or cluster共用的機制
可以做簡單的資料操作,如取符合條件的子集合資料出來
配合其他 big 系列的套件如 biganalytics, bigtabultae等做其他處理、modeling
3. 套件主要函數列表:
a. read.big.matrix: 讀取一份.csv 並創建成file-backing形式的big.matrix
格式比如
read.big.matrix("test.csv", header = TRUE,
type = "double",
backingfile = "test.bin",
descriptorfile = "test.des"))
你提供test.csv, 執行後會多兩個供bigmemory使用的descriptorfile .bin, .des
b. attach.big.matrix: 讀取一份 file-backing big.matrix的descriptor file,
提供套件可以抓到這個 big.matrix object所需的資訊
c. mwhich: 如同base所提供的which,可以對各欄位做篩選
d. write.big.matrix: 將 big.matrix object寫入 file
4. 分享內容:
之前看一些朋友發問有較大容量資料要吃進來,而絕大部分都可以由data.table套件的
fread解決。
bigmemory處理資料當然沒有data.table又快又方便,但它有個好處,就是一開始只放
資料的記憶體指標,不會把所有資料都放進記憶體。
所以我把它應用在網路server上需要供人查詢的較大筆資料(如shiny建構的查詢介面)
資料本身較少更動,而供公眾使用的linux server資源不多(有時VM只開4GB)
當我把資料備妥(.csv),先建好file-backing方式所需要的descriptor file,
之後只要attach上去,資料就可以在web-based application中讀取到。
使用者以介面查詢、篩選資料範圍,透過 mwhich 方式縮小真正載入記憶體的資料大小,
再轉換到data.table做其他運算。
所以我用bigmemory的方式、函數超簡單:attach %>% mwhich (%>% data.table())
這也能用資料庫完成,但上述流程可能比 (connect Database -> SQL query -> return
query)來得快一點(後有簡單測試)
但如果資料本身常常更新,或資料各欄位型態複雜,資料庫有它難以取代的優勢。
bigmemory也可以 write, flush,但我本身很少用它,我主要應用在一份很大的歷史資料
(數值資料,少更動),這當然僅只是個人選擇。
bigmemory另一個不錯的優點也在於它和Rcpp(, RcppArmadillo..)等的配合,比如這個
簡單清楚的例子
http://www.r-bloggers.com/using-rcpparmadillo-with-bigmemory/
其他應用或參考資料 可在R-blogger上搜尋 bigmemory
另外google 這份文件也頗有參考價值,雖然已是2010年...
Taking R to the Limit, Part II: Working with Large Datasets
我之前曾對data.table, bigmemory, 和PostgreSQL做簡單的測試,以下是部分內容(可run
library(data.table)
library(magrittr)
library(bigmemory)
library(RPostgreSQL)
library(microbenchmark)
# ========================== Test data preparation
tstL <- 1e6
wr.first <- TRUE
bigmf<- file("bigm_sample01.csv", open = "w")
DT <- data.table(lat=numeric(), lng=numeric(), date=as.Date(character()),
grp=numeric(), pick=numeric())
idxf <- function(x, idx) {
x[-idx] <- 0; x[idx] <- 1; return(x)
}
pconn <- dbConnect(dbDriver("PostgreSQL"), # change to your configuration
user="xx", password="xx", dbname="xxx", host="localhost")
print("write big.mat start")
print(format(Sys.time(), "%Y%m%d %H:%M:%S"))
# randomly prepare data, can arbitrarily chang loop iteration
for (i in 1:20) {
dt <- data.table(lat = runif(tstL,0,90),lng = runif(tstL,0,180),
yr = as.integer(runif(tstL,2000,2015)),
mo = as.integer(runif(tstL,1,12)),
day = as.integer(runif(tstL,1,28))) %>%
.[,date:=as.Date(paste(yr,mo,day,sep=" "),"%Y %m %d")] %>%
.[,c("yr","mo","day","grp"):=list(NULL,NULL,NULL,i)] %>% setkey(date) %>%
.[,pick:=idxf(seq_along(lat),sample(seq_along(lat),1)), by=.(date)]
print(format(Sys.time(), "%Y%m%d %H:%M:%S"))
print("combine DT")
DT <- rbindlist(list(DT, dt))
print(format(Sys.time(), "%Y%m%d %H:%M:%S"))
print("write to PostgreSQL")
dbWriteTable(pconn, value=dt, name= "bigmdb", append=!wr.first, row.names=F)
print(format(Sys.time(), "%Y%m%d %H:%M:%S"))
print("write.table")
# Big.matrix cannot have mixed-type data, change charater 'date' to int 'datei'
write.table(dt[,datei:=as.integer(gsub("-","",date))] %>% .[,date:=NULL],
file = bigmf, sep = ",", row.names = FALSE, col.names = wr.first)
print(i)
wr.first <- FALSE
}
close(bigmf)
#========================== ? read.big.matrix
system.time(db <- read.big.matrix("bigm_sample01.csv", header = TRUE,
type = "double",
backingfile = "bigm_sample01.bin",
descriptorfile = "bigm_sample01.des"))
# user system elapsed
# 69.21 3.21 72.54
#========================== ? attach.big.matrix
system.time(db <- dget("bigm_sample01.des") %>% attach.big.matrix())
# user system elapsed
# 0.01 0.00 0.01
nrow(db) ## 2e+07 rows
nrow(DT)
#========================== Indexing PostgreSQL data
dbSendQuery(pconn, "CREATE INDEX date_index ON bigmdb USING btree (date)")
#========================== simple benchmark
microbenchmark(
'DT' = DT[date>='2003-01-01' & date <='2014-12-01' & pick==1,],
'Bigm' = db[mwhich(db,c(5,5,4),list(c(20030101,20141201,1)),
list(c('ge','le','eq')),'AND'),],
'SQL'= dbGetQuery(pconn, statement=paste0("SELECT * FROM bigmdb WHERE date >=
'2003-01-01' AND date <= '20141201'
AND pick = 1;")),
times=10
) ######## Note: SQL statement should be in single line ######################
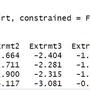
#Unit: milliseconds
#expr min lq mean median uq max neval
# DT 325.0868 347.0721 367.5553 360.3873 389.9555 440.2354 10
#Bigm 404.6935 416.9812 452.5594 441.7622 462.6059 591.9303 10
# SQL 3221.8961 3226.8496 3303.9357 3274.3635 3377.8906 3521.6359 10
format(object.size(DT),"Mb")
#[1] "762.9 Mb"
format(object.size(out2),"Mb")
#[1] "2.7 Mb"
lapply(dbListConnections(PostgreSQL()), dbDisconnect)
5. 備註
沒有特別分享到什麼bigmemory套件高深功能,也不是來騙錢的XDD 只是拋磚引玉,
自己也有困惑~~ 也許data欄位不能mixed-type 某方面侷限了它的發展,bigmemory
在網路的討論度很低,但套件作者默默在維持、不時小更新,只是未來的發展走向不明。
不知道它在愈來愈多更快、更方便的資料處理套件選擇下,未來性如何??~~
bigmemory只是R資料處理中的其一選擇,小小心得供參,也請多多指教,更希望引來
有趣的其他應用或使用方式,謝謝
--
※ 發信站: 批踢踢實業坊(ptt.cc), 來自: 140.112.65.48
※ 文章網址: https://www.ptt.cc/bbs/R_Language/M.1469160184.A.266.html
推
07/22 17:14, , 1F
07/22 17:14, 1F
→
07/22 19:39, , 2F
07/22 19:39, 2F
推
07/22 19:50, , 3F
07/22 19:50, 3F
推
07/22 20:21, , 4F
07/22 20:21, 4F
推
07/23 00:14, , 5F
07/23 00:14, 5F
推
07/23 04:33, , 6F
07/23 04:33, 6F
推
07/23 20:28, , 7F
07/23 20:28, 7F
推
07/24 20:51, , 8F
07/24 20:51, 8F
推
07/24 21:20, , 9F
07/24 21:20, 9F
→
07/24 22:36, , 10F
07/24 22:36, 10F
推
07/24 22:39, , 11F
07/24 22:39, 11F
→
07/24 22:40, , 12F
07/24 22:40, 12F
→
07/24 22:40, , 13F
07/24 22:40, 13F
→
07/24 22:41, , 14F
07/24 22:41, 14F
→
07/24 22:43, , 15F
07/24 22:43, 15F
→
07/24 22:43, , 16F
07/24 22:43, 16F
→
07/24 22:44, , 17F
07/24 22:44, 17F
推
07/24 22:45, , 18F
07/24 22:45, 18F
→
07/24 22:46, , 19F
07/24 22:46, 19F
→
07/24 22:46, , 20F
07/24 22:46, 20F
→
07/24 22:46, , 21F
07/24 22:46, 21F
→
07/24 22:46, , 22F
07/24 22:46, 22F
→
07/24 22:48, , 23F
07/24 22:48, 23F
→
07/24 22:50, , 24F
07/24 22:50, 24F
→
07/24 22:50, , 25F
07/24 22:50, 25F
→
07/24 22:53, , 26F
07/24 22:53, 26F
→
07/24 22:55, , 27F
07/24 22:55, 27F
→
07/24 22:58, , 28F
07/24 22:58, 28F
推
07/24 23:29, , 29F
07/24 23:29, 29F
推
07/24 23:42, , 30F
07/24 23:42, 30F
→
07/24 23:44, , 31F
07/24 23:44, 31F
→
07/24 23:45, , 32F
07/24 23:45, 32F
→
07/24 23:45, , 33F
07/24 23:45, 33F
→
07/25 09:10, , 34F
07/25 09:10, 34F
→
07/25 09:11, , 35F
07/25 09:11, 35F
→
07/25 09:12, , 36F
07/25 09:12, 36F
R_Language 近期熱門文章
PTT數位生活區 即時熱門文章
5
16