[問題] 晶片微陣列程式解釋
ctrl + y 可以刪除一整行,請將不需要的內容刪除
文章分類提示:
- 問題: 當你想要問問題時,請使用這個類別
- 分享: 當你看到別人的心得時,請使用這個類別。版主鼓勵你幫版友歸納重點(選擇性
)
- 情報: 當你看到消息時,請使用這個類別。版主鼓勵你幫版友歸納重點(選擇性)
- 心得: 當你自己想要分享經驗時,請使用這個類別。
- 討論: 當你自己已經有答案,但是也想聽聽版友意見時
問題
[問題類型]:程式諮詢
意見調查(我對R 有個很棒的想法,想問問大家的意見)
程式諮詢(我想用R 做某件事情,但是我不知道要怎麼用R 寫出來)
效能諮詢(我想讓R 跑更快)
經驗諮詢(我想用R 連接某些資料庫,請問大家的經驗)
程式諮詢
[軟體熟悉度]:入門
請把以下不需要的部份刪除
新手(沒寫過程式,R 是我的第一次)
入門(寫過其他程式,只是對語法不熟悉)
使用者(已經有用R 做過不少作品)
開發者(有撰寫R 的套件經驗)新手
[問題敘述]:由於不是很懂R,麻煩懂的人幫我解釋一下最下面那段程式碼(每一行的設計意義),晶片的位置在這邊下載(http://www.ebi.ac.uk/arrayexpress/files/E-MEXP-569/E-MEXP-569.raw.1.zip)
請簡略描述你所要做的事情,或是這個程式的目的
[程式範例]:source("http://bioconductor.org/biocLite.R")
biocLite("affy")
biocLite("GeneNet")
biocLite("limma")
biocLite("tkWidgets")
biocLite("Biobase")
biocLite("AffyID2GeneID")
source("http://faculty.ucr.edu/~tgirke/Documents/R_BioCond/My_R_Scripts/GOHyperGAll.txt")
AffyID2GeneID(map =
"ftp://ftp.arabidopsis.org/home/tair/Microarrays/Affymetrix/affy_ATH1_array_elements-2010-12-20.txt")
library(affy)
library(limma)
library(tkWidgets)
library(annaffy)
library(GeneNet)
------------
P_syr_avr<-ReadAffy(widget=TRUE)
-------------------------------
rma_P_syr_avr<-rma(P_syr_avr)
------------------------------
rep.day <- rep(c("A","B"),4)
times <- sort(gl(4,2))
design <- model.matrix(~factor(times) + factor(rep.day))
colnames(design) <- c(as.character(1:4),"B")
cont.matrix <- rbind(0,diag(3),0)
-------------------------------
fit <- lmFit(rma_P_syr_avr,design)
rma <- rma_P_syr_avr
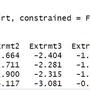
-----------------f-p-value 值 ---DEG
fit2 <- contrasts.fit(fit, cont.matrix)
fit2 <- eBayes(fit2)
modF001 <- fit2$F.p.value[fit2$F.p.value<0.01]
modF025 <- fit2$F.p.value[fit2$F.p.value<0.025]
modF <- fit2$F.p.value
o <- order(modF)
which.A <- seq(1,7,2)
which.B <- seq(2,8,2)
x=cbind(rownames(exprs(rma)),fit2$F.p.value)
write(t(x),"d:/x.txt",ncol=ncol(x),sep="\t")
-----------------f-p-value 值
x<-c()
count_i = length(modF001)
for(count_j in 1: (count_i-1)){
for(count_k in (count_j+1): count_i){
pcc <- cor(exprs(rma)[o[count_j],which.A],exprs(rma)[o[count_k],which.A])
abs_pcc <- abs(pcc)
if(abs_pcc>0.95){
x1<-c(AffyID2GeneID(affyIDs=rownames(exprs(rma))[o[count_j]]),
AffyID2GeneID(affyIDs=rownames(exprs(rma))[o[count_k]]), pcc)
x<-rbind(x,x1)
}
}
}
write(t(x),"d:/481.txt",ncol=ncol(x),sep="\t")
-
---------------------------請幫忙解釋以下這段--------------------------------------------
data<-read.table("d:/x2.txt",sep="\t")
head(exprs(rma))
probe1=as.character(data$V1)
exprs(rma)[rownames(exprs(rma))==probe1[3]]
x_481=numeric()
count_i = length(probe1)
for(count_j in 1: count_i){
x_481 =rbind(x_481,c(exprs(rma)[rownames(exprs(rma))==probe1[count_j]]))
}
rownames(x_481)=probel[1:count_i]
library(GeneNet)
pcor.dyn <- ggm.estimate.pcor(t(x_481), method = "dynamic")
arth.edges <- network.test.edges(pcor.dyn,direct=TRUE)
arth.net <- extract.network(arth.edges, method.ggm="number", cutoff.ggm=1200)
nodePP=cbind(arth.net[,1],as.character(probe1[arth.net[,2]]),as.character(probe1[arth.net[,3]]))
write.table(nodePP,"d:/481.txt")
張貼能夠重現錯誤的程式碼,可以幫助版友更快的幫你解決問題
程式碼可貼於以下網站:
http://ideone.com/
http://codepad.org
http://pastie.org/
http://nopaste.info/
http://pastebin.com/
http://paste.plurk.com
http://gist.github.com/
http://nopaste.csie.org/
[關鍵字]:
選擇性,也許未來有用
--
※ 發信站: 批踢踢實業坊(ptt.cc), 來自: 122.118.21.180
※ 文章網址: http://www.ptt.cc/bbs/R_Language/M.1400316155.A.D46.html
→
05/17 17:20, , 1F
05/17 17:20, 1F
→
05/17 17:32, , 2F
05/17 17:32, 2F
→
05/17 17:33, , 3F
05/17 17:33, 3F
R_Language 近期熱門文章
PTT數位生活區 即時熱門文章