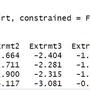
[問題] 以R軟體解ODE (傳染病數學模型)
[問題類型]:
程式諮詢(我想用R 做某件事情,但是我不知道要怎麼用R 寫出來)
[軟體熟悉度]:
入門(寫過其他程式,只是對語法不熟悉)
[問題敘述]:
某傳染病流行病學課本的公式:
n: size of the population
S: proportion of n that is susceptible
I: proportion of n that is currently infected and infectious
R: proportion of n that is immune.
D: 從感染到痊癒所需的時間
β:接觸患者後感染的機率
κ:單位時間內平均一個人會接觸幾個人
S_t+1 = S_t - βκS_tI_t
I_t+1 = I_t + βκS_tIt - I_t/D
R_t+1 = R_t + I_t/D
(↑這些叫做差分方程,對吧?)
dS/dt = - βκS_tI_t
dI/dt = βκS_tI_t - I_t/D
dR/dt = I_t/D
(↑要解出給定時間的S,I,R,應該就是解微分方程?)
請問如果要把dS/dt, dI/dt, dR/dt在同一張圖上作圖,該怎麼做?
另外還有S, I, R對時間作圖。
最主要是想了解dI/dt是否會在某段時間之後趨近於0。
以下程式執行結果跟預期的差很多...
不知道是不是function沒寫對?
[程式範例]:
課本給的參數是beta=0.15(/人), kapp=12(人/週), D=1(週), 時間單位是週
初始條件:族群總人數為1000人,有1人感染。
http://pastie.org/7682058
library(deSolve)
pars <- c(beta=0.15, kappa=12, D=1, pop_size=1000)
yini <- c(I=1, S=999, R=0)
times <- seq(0, 25, by=1)
infection <- function(Time, State, Pars) {
with(as.list(c(State, Pars)), {
dS <- -beta*kappa*(S/pop_size)*(I/pop_size)
dI <- beta*kappa*(S/pop_size)*(I/pop_size)-((I/pop_size)/D)
dR <- (I/pop_size)/D
return(list(c(dS, dI, dR)))
})
}
out<- ode(func=infection,y=yini,parms=pars,times=times)
matplot(out[,"time"], out[,2:4], type = "l", xlab = "week", ylab = "Rate",
main = "Infection", lwd = 2)
legend("topright", c("dS/dt", "dI/dt", "dR/dt"), col = 1:3, lty = 1:2)
[關鍵字]:
R, ODE, infection epidemiology, mathematical model
PS:
我自己用excel做的簡易計算結果在這邊(主要是要看dI/dt對時間作圖的粗略結果)
,可以參考看看。
http://www.filethief.com/download/1983/epi_math.xlsx.html
感謝指教!
--
※ 發信站: 批踢踢實業坊(ptt.cc)
◆ From: 111.255.5.34
→
04/20 13:47, , 1F
04/20 13:47, 1F
→
04/20 13:51, , 2F
04/20 13:51, 2F
推
04/21 11:28, , 3F
04/21 11:28, 3F
→
04/21 11:29, , 4F
04/21 11:29, 4F
→
04/21 11:29, , 5F
04/21 11:29, 5F
→
04/21 11:30, , 6F
04/21 11:30, 6F
→
04/21 11:32, , 7F
04/21 11:32, 7F
→
04/21 11:33, , 8F
04/21 11:33, 8F
感謝各位回答!
剛開始先改yini的順序,但還是執行不出預期結果。
後來參考其他人用R語言實作SIR model的程式,
http://archives.aidanfindlater.com/blog/2010/04/20/the-basic-sir-model-in-r/
發現似乎直接把S,I,R定義成proportion就好...
(也就是要把S,I定成999/1000,1/1000)
(如我上述的S,I,R定義,這三個變數其實是proportion)
我一開始是把S,I,R定義成人數,再讓它除以pop_size(族群總人口數),
dS <- -beta*kappa*(S/pop_size)*(I/pop_size)
dI <- beta*kappa*(S/pop_size)*(I/pop_size)-((I/pop_size)/D)
dR <- (I/pop_size)/D
這樣是不符合SIR model公式的。
以下ODE解出來的SIR都是proportion,
但是最後會把out矩陣中的SIR值再乘上pop_size,
以求出人數。
http://pastie.org/7682055
library(deSolve)
pop_size=1000
pars <- c(beta=0.15, kappa=12, D=1)
yini <- c(S=0.999, I=0.001, R=0)
times <- seq(0, 25, by=1)
infection <- function(Time, State, Pars) {
with(as.list(c(State, Pars)), {
dS <- -beta*kappa*S*I
dI <- beta*kappa*S*I-(I/D)
dR <- I/D
return(list(c(dS, dI, dR)))
})
}
out<- ode(func=infection,y=yini,parms=pars,times=times)
out[,2:4] <- pop_size*out[,2:4]
matplot(out[,"time"], out[,2:4], type = "l", xlab = "week", ylab = "numbers",
main = "Infection", lwd = 2)
legend("topright", c("Susceptibles", "Infecteds", "Recovereds"), col = 1:3,
lty = 1:2)
不過,現在還有個問題是...
怎麼作出dS/dt, dI/dt, dR/dt對時間作圖的結果呢?
因為在這個課本的範例中,
大約在第二十週之後感染者人數幾乎是零人,
而且從SIR的圖也可看出,感染者增加的速率是先上升後下降的。(由切線斜率判斷)
也就是說...我似乎需要先寫出d^2X/dt^2 (X=S,I,R)的function才行?
這下變成純粹的數學問題了= ="
I''= βκ(S'I+SI') –I'/D
這樣寫對嗎?
有辦法用R解這種型態的ODE嗎?
→
04/22 00:37, , 9F
04/22 00:37, 9F
呃...會有影響。
因為這公式對S,I,R的定義就是proportion,
所以要寫成d(S/pop_size)/dt才算得出來?
→
04/22 00:38, , 10F
04/22 00:38, 10F
→
04/22 00:39, , 11F
04/22 00:39, 11F
感謝指教...
可是out[-1,2] - out[-nrow(out),2]無法把times這個column留下來?
out<- ode(func=infection,y=yini,parms=pars,times=times)
out[,2:4] <- pop_size*out[,2:4]
dout<- out[-1,1:4] - out[-nrow(out),1:4]
week <- times[2:26]
dim(week) <-c(25,1)
#因為times都變成1,只好設定一個叫做week的矩陣代表時間
matplot(week, dout[,2:4], type = "l", xlab = "week", ylab = "numbers", main =
"Infection", lwd = 2)
legend("topright", c("dS/dt", "dI/dt", "dR/dt"), col = 1:3, lty = 1:2)
推
04/22 18:47, , 12F
04/22 18:47, 12F
感謝回答!
※ 編輯: anovachen 來自: 111.255.244.92 (04/23 23:15)
R_Language 近期熱門文章
PTT數位生活區 即時熱門文章